BIG-Annotator:基因组测序数据高效功能注释及其在遗传诊断中的应用
Application of BIG-Annotator in the genome sequencing data functional annotation and genetic diagnosis
BIG-Annotator:基因组测序数据高效功能注释及其在遗传诊断中的应用 |
| 黄莹,刘琪,池连江,石承民,吴祯,胡敏,石宏,陈华 |
|
Application of BIG-Annotator in the genome sequencing data functional annotation and genetic diagnosis |
| Huang Ying,Liu Qi,Chi Lianjiang,Shi Chengmin,Wu Zhen,Hu Min,Shi Hong,Chen Hua |
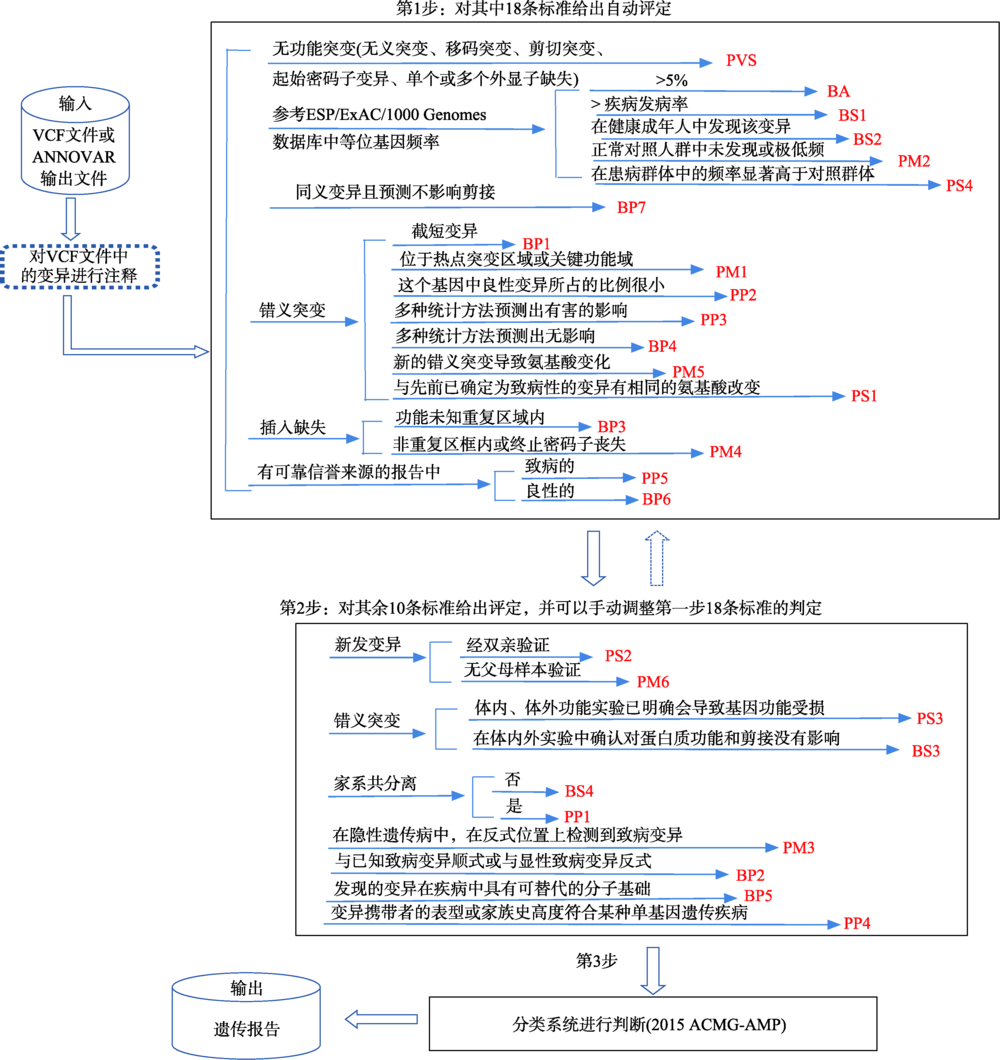
| 图1 BIG-Annotator分析流程 PVS:当一个疾病的致病机制为功能丧失(LOF)时的无功能变异(无义突变、移码突变、经典±1或2的剪接突变、起始密码子变异、单个或多个外显子缺失)。PM1:位于热点突变区域,和/或位于已知无良性变异的关键功能域(如酶的活性位点)。PM4:非重复区框内插入/缺失或终止密码子丧失导致的蛋白质长度变化。PM5:新的错义突变到氨基酸变化,此变异之前未曾报道,但是在同一位点,导致另外一种氨基酸的变异已经确认是致病性的。PP2:对某个基因来说,如果这个基因的错义变异是造成某种疾病的原因,并且这个基因中良性变异所占的比例很小,在这样的基因中所发现的新的错义变异。PP3:多种统计方法预测出该变异会对基因或基因产物造成有害的影响,包括保守性预测、进化预测、剪接位点影响等。PP5:有可靠信誉来源的报告认为该变异为致病的,但证据尚不足以支持进行实验室独立评估。BS2:对于早期完全外显的疾病,在健康成年人中发现该变异(隐性遗传病发现纯合、显性遗传病发现杂合,或者X连锁半合子)。BP1:已知一个疾病的致病原因是由于某基因的截短变异,在此基因中所发现的错义变异。BP2:在显性遗传病中又发现了另一条染色体上同一基因的一个已知致病变异,或者是任意遗传模式遗传病中又发现了同一条染色体上同一基因的一个已知致病变异。BP3:功能未知重复区域内的缺失/插入,同时没有导致基因编码框改变。BP4:多种统计方法预测出该变异会对基因或基因产物无影响,包括保守性预测、进化预测、剪接位点影响等。注意事项:由于做预测时许多生物信息算法使用相同或非常相似的输入,每个算法不应该算作一个独立的标准。BP4在一个任何变异的评估中只能使用一次。BP6:有可靠信誉来源的报告认为该变异为良性的,但证据尚不足以支持进行实验室独立评估。 |
| Fig. 1 Pipeline of BIG-Annotator |

|