水稻典型品种日本晴和IR24根系微生物组的解析
Analysis of rice root bacterial microbiota of Nipponbare and IR24
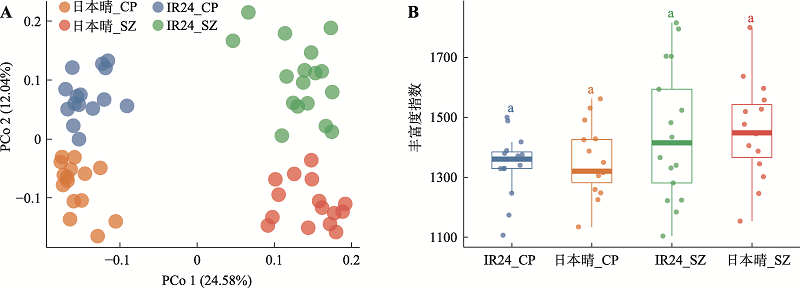
A:基于样品间Bray Curtis距离的主坐标分析(PCoA)。日本晴和IR24的根系微生物组在PCoA的第一轴上(PCo 1)按种植地点开,第二轴上(PCo 2)按基因型分开,组间差异显著(PERMANOVA, P < 0.001)。CP:昌平;SZ:上庄;坐标轴标题括号中显示整体差异的解释率百分比。B:日本晴和IR24根系微生物组的丰富度指数(richness index,样品内物种多样性)。箱线图中框内横线表示中位数,上下边缘分别代表上下四分位数,边缘上的延长线在无异常值时至极值,但最长不超过1.5倍上下四分位数的分布区间。各组间丰富度指数差异不明显,最小显著差异法(least significant difference, LSD)比较组间差异,4组均为a表示地点和基因型对根系微生物组的Alpha多样性影响不显著(P > 0.05)。