水稻典型品种日本晴和IR24根系微生物组的解析
Analysis of rice root bacterial microbiota of Nipponbare and IR24
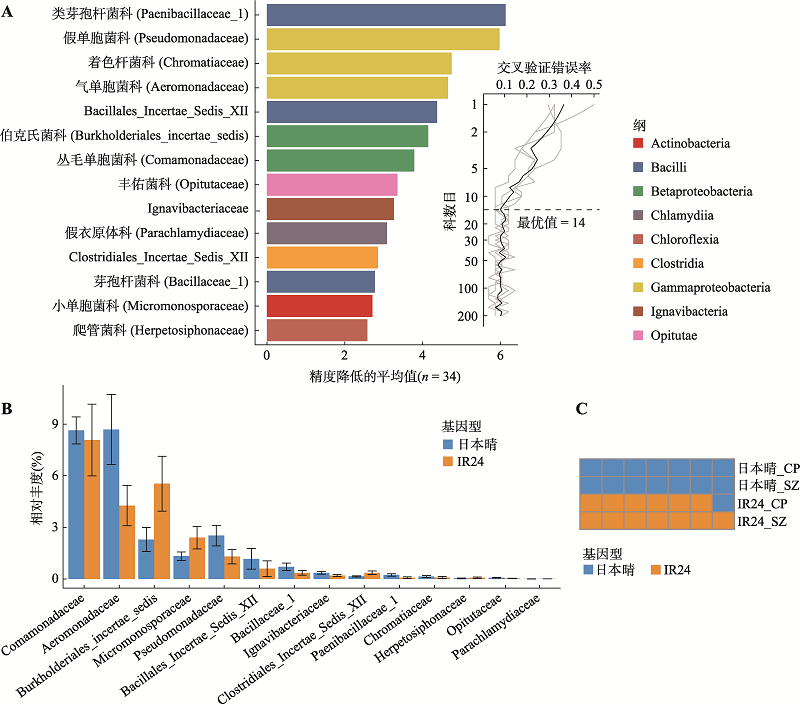
A:随机森林分类模型中重要性排前14的生物标记菌。采用随机森林分类法,对昌平和上庄地区的IR24和日本晴根系微生物组(共34个样品)在科水平进行了分类,并在纲水平着色。生物标记菌按照对模型准确度的重要性由高到低排序,展示前14个。图右侧小图表示十倍交叉验证错误率,当标记菌个数超过14个时,模型的错误率较低且稳定。B:标记菌在日本晴和IR24根系微生物组中的相对丰度。蓝色为日本晴,橙色为IR24。C:用随机森林分类模型对两个基因型做分类预测。该模型基于样品的根系微生物组成判断样品的基因型。前两行样品为日本晴,后两行样品为IR24,蓝色表示样品基因型被预测为日本晴,橙色则是预测为IR24。CP:昌平;SZ:上庄。Actinobacteria:放线菌纲;Bacilli:杆菌纲;Betaproteobacteria:beta-变形菌纲;Chlamydiia:衣原体纲;Chloroflexia:绿弯菌纲;Clostridia:梭菌纲;Gammaproteobacteria:gamma-变形菌纲;Opitutae:丰佑菌纲。