Hereditas(Beijing) ›› 2025, Vol. 47 ›› Issue (2): 271-285.doi: 10.16288/j.yczz.24-254
• Review • Previous Articles
A new era of evolutionary analysis based on a personal computer: the future of multifunctional software such as eGPS
Dalang Yu1,3( ), Jianing Yang2,3, Jianwei Zhang2,3, Wanyu Zhang2,3, Haipeng Li2,3(
), Jianing Yang2,3, Jianwei Zhang2,3, Wanyu Zhang2,3, Haipeng Li2,3( )
)
- 1. Key Laboratory of Systems Health Science of Zhejiang Province, School of Life Science, Hangzhou Institute for Advanced Study, Hangzhou 310024, China
2. CAS Key Laboratory of Computational Biology, Shanghai Institute of Nutrition and Health, Shanghai 200031, China
3. University of Chinese Academy of Sciences, Beijing 100049, China
-
Received:2024-09-03Revised:2024-11-08Online:2025-02-20Published:2024-11-27 -
Contact:Haipeng Li E-mail:yudalang@ucas.edu.cn;lihaipeng@sinh.ac.cn -
Supported by:Youth Fund of the National Natural Science Foundation of China(32400511)
Cite this article
Dalang Yu, Jianing Yang, Jianwei Zhang, Wanyu Zhang, Haipeng Li. A new era of evolutionary analysis based on a personal computer: the future of multifunctional software such as eGPS[J]. Hereditas(Beijing), 2025, 47(2): 271-285.
share this article
Add to citation manager EndNote|Reference Manager|ProCite|BibTeX|RefWorks
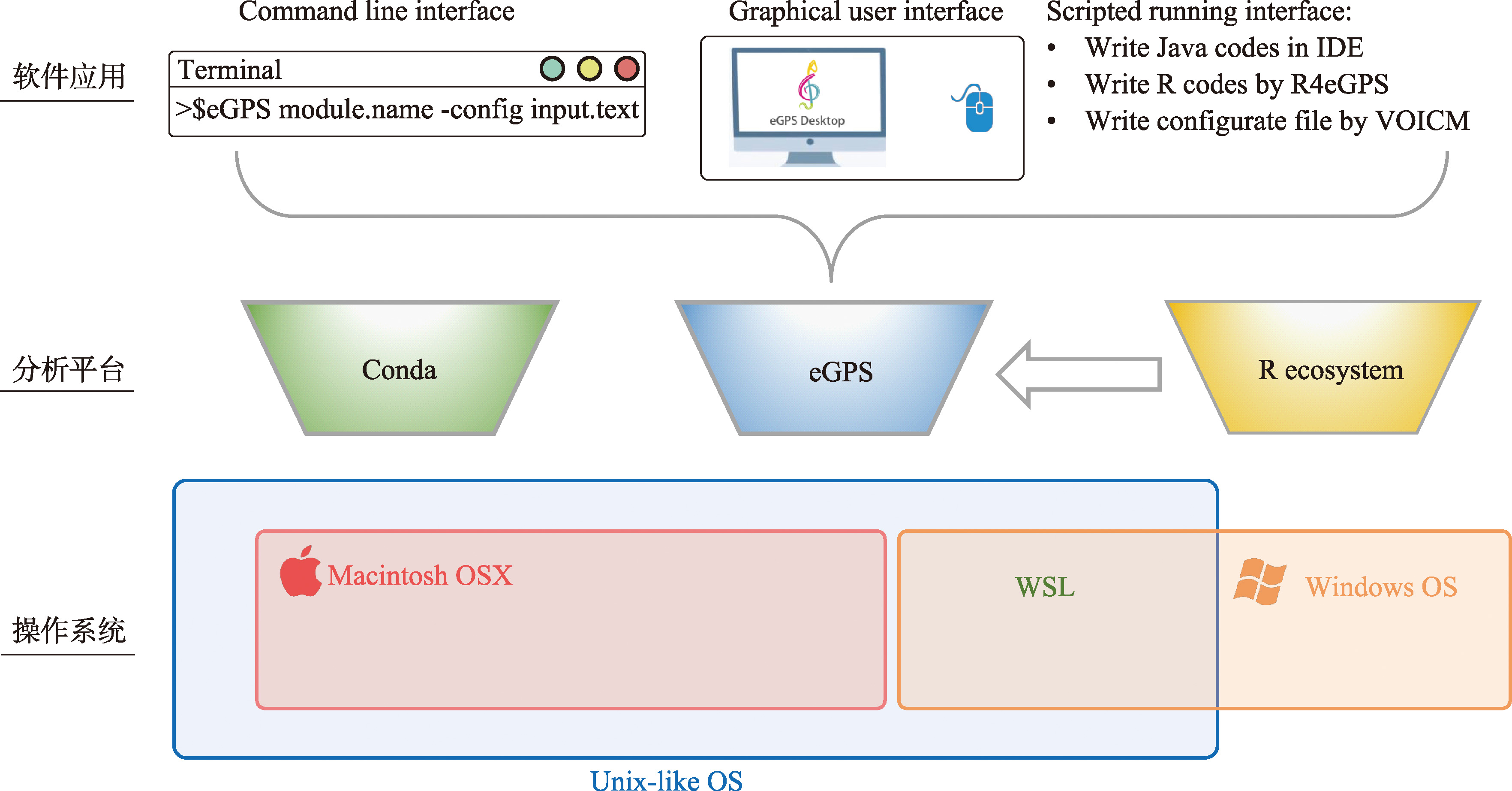
Table 1
Comparison of bioinformatics analysis using personal computers, cloud-based data analysis, and high-performance computing clusters"
| 数据分析途径 | PC机 | 云平台 | 高性能计算集群 |
|---|---|---|---|
| 运行方式 | 下载安装软件后使用,可通过图形界面、命令行与脚本运行 | 用户打开浏览器使用 | 用户主要通过终端软件命令行操作,也可以通过浏览器访问,或者通过图形界面 |
| 获取难易程度 | 容易 | 普通,需向商家购买服务 | 困难,需配备机房和相关设备,需要专人维护 |
| 功能可达性 | 需要下载软件 | 需要联网 | 需要局域网 |
| 用户更新软件 难易程度 | 需用户重新下载软件或软件提供增量更新方式。 | 无需用户主动更新 | 需要集群联网或用户手动下载软件更新 |
| 用户使用软件 上手难度 | 依赖于使用方式,图形界面软件较方便,命令行软件有一定使用成本,脚本化使用方式软件成本更高 | 容易 | 有一定学习成本 |
| 操作延迟性 | 几乎无延迟 | 依赖网络带宽与平台性能 | 依赖局域网带宽 |
| 数据安全性 | 仅依赖个人 | 依赖个人、服务商 | 依赖个人、单位 |
| 服务稳定性 | 稳定 | 若平台关闭服务则无法使用 | 取决于研究机构的情况 |
| 部署的难易程度 | 容易 | 需要专人部署、专人维护 | 需要专人部署、专人维护 |
| 开发成本 | 个人即可完成开发,一般仅需一门编程语言 | 需团队分工,多门语言配合 | 个人即可完成开发,除基本编程语言外,需掌握shell脚本与vim编辑器的使用 |
| 开发周期 | 开发周期短,开发者更新完成即可编译、发布 | 开发周期长,开发完备后需要重新部署、并重启,需要考虑周到 | 开发周期短,开发者更新完成即可编译、发布 |
| 开发便利性 | 开发方便,可使用集成开发环境(IDE) | 依赖云平台是否支持开发,便利性取决于云平台是否提供网页版IDE | 开发较方便,可安装JupterLab、Rstudio Server等工具提升开发便利性 |
| 内存限制 | 新购置的常规PC机一般为16 G,内存容量仍在不断增大 | 取决于提供服务的商家 | 内存一般达TB级别 |
| 硬盘情况 | 固态硬盘不断流行,还可外接硬盘 | 取决于提供服务的商家 | 可获得TB级别存储空间 |
| 功能多样性 | 多样 | 取决于平台是否支持开发与自主安装软件 | 多样 |
| 计算速度 | 取决于PC机性能 | 取决于平台商业策略 | 取决于使用的服务器的性能 |
| 是否经济 | 是 | 否,一般试用期免费 | 否 |
Table 2
Representative software for bioinformatics analysis on PC and their main features"
| 特性 | 代表软件 | ||||||
|---|---|---|---|---|---|---|---|
| Microsoft Excel | MEGA | DnaSP | Cytoscape | Galaxy/UGENE | DNASTAR/ Geneious | eGPS | |
| 研究领域 | 通用数据分析 | 传统进化领域 | 群体遗传学 | 系统生物学 | 数据分析平台 | 综合序列分析软件 | 数据分析平台 |
| 官方网址 | |||||||
| 主要功能 | 数据记录、统计分析、作图 | 序列分析、统计计算、可视化 | 遗传多样性、中性检验 | 可视化分子相互作用网络和生物通路 | 可视化、分析流程工具整合 | 序列分析、分析流程整合等 | 进化分析、可视化与小工具等 |
| 个人是否免费使用 | 否 | 是 | 是 | 是 | 是 | 否 | 是 |
| 部署的难易程度 | 容易 | 容易 | 容易 | 容易 | Galaxy困难 UGENE容易 | 容易 | 容易 |
| 是否支持开发插件 | 是 | 否 | 否 | 是 | 是 | 否 | 是 |
| 最新版本(更新时间) | Excel 2021 (2024.09) | 11.0.13 (2022.06) | 6.12.03 (2019.02) | 3.10.2 (2024) | 24.1(2024.06)/ 51.0(2024.09) | NA / 2024.0 | 2.0.4.12 (2024.09) |
| 运行方式 | GUI/ Script | GUI/ CL | GUI | GUI/Web | Web/GUI | GUI | GUI/CL/ Script |
| 是否公布脚本化运行API | 是(VBA编程) | 否 | 否 | 是(Java编程) | 是(JavaScript, Python等) | 否 | 是(Java编程) |
| 软件创新点 | 与Window操作系统融合度高 | 开发早、分析方法全、提供图注 | 开发早、提供GUI | 复杂网络分析和可视化的通用平台 | 功能多样;贴合用户 | 功能多样;商业团队支持 | 进化与多组学融合;支持大数据分析、提供大型可视化模块 |
Table 3
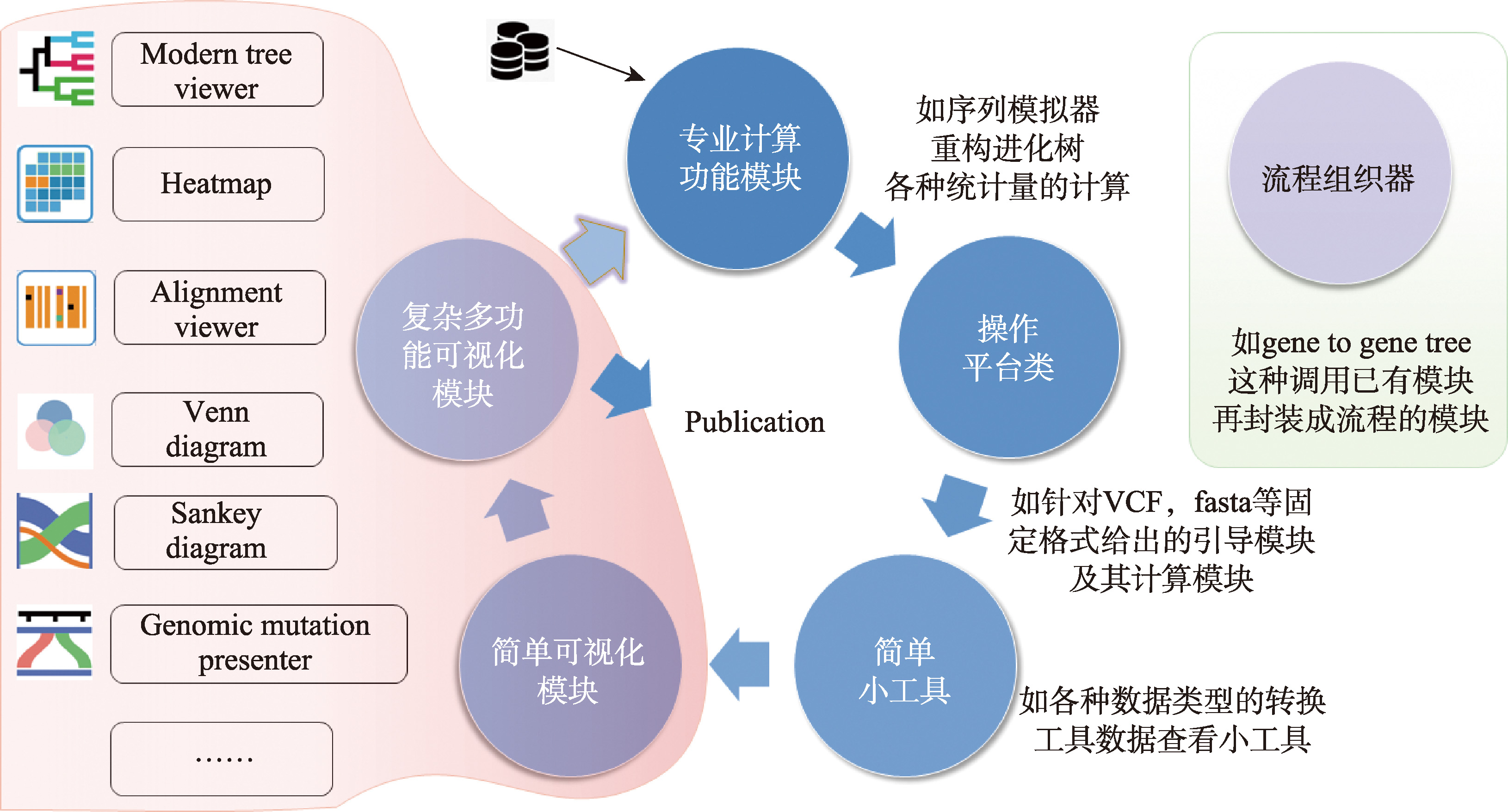
Functional module list of eGPS"
| 模块英文名 | 模块中文名 | 注备 | 进化分析类型 |
|---|---|---|---|
| Gene family browser | 基因家族浏览器 | 展示不同物种中的通路情况,用户能够以交互式的 方式展示进化树 | 通路 |
| Heatmap | 热图 | 通用基础可视化 | 基因 |
| Modern tree view | 现代进化树展示器 | 友好且高度交互式的进化树展示、编辑与操作 | 基因、基因组 |
| Venn plot | 韦恩图 | 通用基础可视化 | 通用 |
| Multi-seqs struct view | 多序列结构展示器 | 多基因序列结构的展示器 | 基因 |
| Pathway illuminator | 通路展示器 | 通路情况的绘制图 | 通路 |
| Mutation presenter | 突变展示器 | 序列突变情况展示器,可关注保守区域与多态区域 | 基因 |
| Sanky plot | 桑基图 | 通用基础可视化以及序列演化展示 | 通用 |
| Alignment view | 多序列联配展示器 | 联配展示器 | 基因、基因组 |
| Gene browser | 基因结构浏览器 | 方便地展示基因结构 | 基因 |
| Bed merger | Bed文件合并器 | 中间过程文件操作器 | 通用 |
| Tree tanglegram | 纠缠图 | 两个进化树比较器 | 基因、基因组 |
| Tree heatmap and gene struct | 树热图和基因结构视图 | 进化树、热图和基因结构展示器 | 基因 |
| String set operator | 字符集合操作器 | 代表小工具 | 通用 |
| Two strings comparator | 双字符串比较器 | 双序列快速展示图 | 基因 |
| Tree Leaf Info Collector | 进化树叶子收集器 | 代表性进化树快速操作工具 | 基因、基因组 |
| Fasta dumper | Fasta倾泻器 | 序列操作工具 | 基因、通路 |
| Fasta Tools | Fasta工具包 | 序列文件快捷操作器 | 基因、通路 |
| GFF3 operator | GFF3操作器 | 基因组注释文件快捷操作器 | 通路、基因组 |
| Quick histogram | 快速直方图 | 通用统计小工具 | 统计小工具 |
| Large volume text view | 大文本编辑器 | 通用计算机工具 | 基因组 |
| Low volume text view | 小文本编辑器 | 通用计算机工具,可支持大规模数据 | 通用 |
| Local blast wrapper | 本地Blast | 一个Blast快速使用的Wrapper | 基因、通路 |
| Mutation string presenter | 突变字符串展示器 | 快速查看序列联配情况 | 基因 |
| Gene to MSA | 基因到联配 | 快速从公共数据库获取基因联配 | 基因 |
| Multi-sequences aligner: MAFFT | 多序列联配包装器 | eGPS软件唯一集成的专业工具 | 基因 |
| Quick reference-based aligner | 基于参考基因比联配器 | 序列联配 | 基因 |
| Alignment string diversity | 联配多样性描述器 | 快速查看序列联配情况 | 基因 |
| Alignment trimmer | 联配修边器 | 序列联配质控 | 基因 |
| Evolutionary dist view | 进化距离展示器 | 可视化进化距离矩阵 | 基因、通路、基因组 |
| Distance calculator: from MSA | MSA到进化距离计算器 | 查看进化距离 | 基因、通路 |
| Distance calculator: from MAF | MAF到进化距离计算器 | 查看进化距离 | 基因组 |
| Gene to evolutionary distance | 基因到进化距离 | 查看进化距离 | 基因 |
| Tree builder: from MSA | 从MSA构建进化树 | 构建进化树 | 基因、通路 |
| Tree builder: from VCF | 从VCF构建进化树 | 构建进化树 | 基因组 |
| Tree builder: from distance | 从距离构建进化树 | 从进化距离构建进化树流程 | 基因、通路、基因组 |
| Gene to gene tree | 基因到基因树 | 构建进化树的快速流程,可初步查看粗略查看基因树 | 基因 |
| Sequence logo | 序列Logo展示器 | 查看序列的基序 | 基因 |
| Classic skyline plot | 传统天际图 | 传统方式估计群体历史 | 基因组 |
| Stairway plot[ | Stairway图 | 估计群体历史 | 基因组 |
| FitCoal[ | 飞得酷 | 估计群体历史 | 基因组 |
| [1] | Eisenstein M. Seven technologies to watch in 2024. Nature, 2024, 625(7996): 844-848. |
| [2] | Yang JN, Cui YM, Yu DL, Zhang GQ, Cao RF, Gu ZL, Dai GY, Wu XX, Ling YC, Yi CY, Sun XY, Sun B, Lin X, Zhang Y, Zhao GP, Li YX, Pan YH, Li HP. A noncoding A-to-U Kozak site change related to the high transmissibility of Alpha, Delta, and Omicron VOCs. Mol Biol Evol, 2023, 40(6): msad142. |
| [3] |
Tamura K, Stecher G, Kumar S. MEGA11: molecular evolutionary genetics analysis version 11. Mol Biol Evol, 2021, 38(7): 3022-3027.
doi: 10.1093/molbev/msab120 pmid: 33892491 |
| [4] |
Xia XH. DAMBE7: new and improved tools for data analysis in Molecular Biology and Evolution. Mol Biol Evol, 2018, 35(6): 1550-1552.
doi: 10.1093/molbev/msy073 pmid: 29669107 |
| [5] |
Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res, 2003, 13(11): 2498-2504.
doi: 10.1101/gr.1239303 pmid: 14597658 |
| [6] | Yu DL, Dong LL, Yan FQ, Mu HL, Tang BX, Yang X, Zeng T, Zhou Q, Gao F, Wang ZH, Hao ZQ, Kang HG, Zheng Y, Huang HW, Wei YZ, Pan W, Xu YC, Zhu JW, Zhao SL, Wang CR, Wang PY, Dai L, Li MS, Lan L, Wang YW, Chen H, Li YX, Fu YX, Shao Z, Bao YM, Zhao FQ, Chen LN, Zhang GQ, Zhao WM, Li HP. eGPS 1.0: comprehensive software for multi-omic and evolutionary analyses. Natl Sci Rev, 2019, 6(5): 867-869. |
| [7] | Hogeweg P. The roots of bioinformatics in theoretical biology. PLoS Comput Biol, 2011, 7(3): e1002021. |
| [8] | Plotree D, Plotgram D. PHYLIP-Phylogeny Inference Package (Version 3.2). Cladistics, 1989, 5: 163-166. |
| [9] | Blum R, Bresnahan C. Linux Command Line and Shell Scripting Bible. John Wiley & Sons, 2015. |
| [10] | Shneiderman B, Plaisant C. Designing The User Interface: Strategies For Effective Human-computer Interaction. Addison-Wesley, 2010. |
| [11] |
Rozas J, Ferrer-Mata A, Sánchez-DelBarrio JC, Guirao- Rico S, Librado P, Ramos-Onsins SE, Sánchez-Gracia A. DnaSP 6: DNA sequence polymorphism analysis of large data sets. Mol Biol Evol, 2017, 34(12): 3299-3302.
doi: 10.1093/molbev/msx248 pmid: 29029172 |
| [12] |
Excoffier L, Lischer HEL. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Resour, 2010, 10(3): 564-567.
doi: 10.1111/j.1755-0998.2010.02847.x pmid: 21565059 |
| [13] |
Huber W, Carey VJ, Gentleman R, Anders S, Carlson M, Carvalho BS, Bravo HC, Davis S, Gatto L, Girke T, Gottardo R, Hahne F, Hansen KD, Irizarry RA, Lawrence M, Love MI, MacDonald J, Obenchain V, Oleś AK, Pagès H, Reyes A, Shannon P, Smyth GK, Tenenbaum D, Waldron L, Morgan M. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods, 2015, 12(2): 115-121.
doi: 10.1038/nmeth.3252 pmid: 25633503 |
| [14] | Hutchison WJ, Keyes TJ, tidyomics Consortium, Crowell HL, Serizay J, Soneson C, Davis ES, Sato N, Moses L, Tarlinton B, Nahid AA, Kosmac M, Clayssen Q, Yuan V, Mu WC, Park JE, Mamede I, Ryu MH, Axisa PP, Paiz P, Poon CL, Tang M, Gottardo R, Morgan M, Lee SR, Lawrence M, Hicks SC, Nolan GP, Davis KL, Papenfuss AT, Love MI, Mangiola S. The tidyomics ecosystem: enhancing omic data analyses. Nature Methods, 2024, 21(7): 1166-1170. |
| [15] | Yu DL, Yang X, Tang BX, Pan YH, Yang JN, Duan GY, Zhu JW, Hao ZQ, Mu HL, Dai L, Hu WJ, Zhang MC, Cui Y, Jin T, Li CP, Ma L, Language translation team, Su X, Zhang GQ, Zhao WM, Li HP. Coronavirus GenBrowser for monitoring the transmission and evolution of SARS-CoV-2. Brief Bioinform, 2022, 23(2): bbab583. |
| [16] | Gamma E, Helm R, Johnson R, Vlissides J. Design Patterns: Elements of Reusable Object-oriented Software. Addison-Wesley Professional, 1994. |
| [17] | Wickham H. Tidy data. J Stat Softw, 2014, 59(10): 1-23. |
| [18] |
Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Mol Biol Evol, 2013, 30(4): 772-780.
doi: 10.1093/molbev/mst010 pmid: 23329690 |
| [19] |
Minh BQ, Schmidt HA, Chernomor O, Schrempf D, Woodhams MD, von Haeseler A, Lanfear R. IQ-TREE 2: new models and efficient methods for phylogenetic inference in the genomic era. Mol Biol Evol, 2020, 37(5): 1530-1534.
doi: 10.1093/molbev/msaa015 pmid: 32011700 |
| [20] |
Lin K, Li HP, Schlötterer C, Futschik A. Distinguishing positive selection from neutral evolution: boosting the performance of summary statistics. Genetics, 2011, 187(1): 229-244.
doi: 10.1534/genetics.110.122614 pmid: 21041556 |
| [21] | Álvarez-Carretero S, Kapli P, Yang ZH. Beginner's guide on the use of PAML to detect positive selection. Mol Biol Evol, 2023, 40(4): msad041. |
| [22] | Nowogrodzki J. ChatGPT for science: how to talk to your data. Nature, 2024, 631(8022): 924-925. |
| [23] |
Liao Y, Smyth GK, Shi W. featureCounts: an efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics, 2014, 30(7): 923-930.
doi: 10.1093/bioinformatics/btt656 pmid: 24227677 |
| [24] | Chen CJ, Wu Y, Li JW, Wang X, Zeng ZH, Xu J, Liu YL, Feng JT, Chen H, He YH, Xia R. TBtools-II: a. “one for all, all for one” bioinformatics platform for biological big-data mining. Mol Plant, 2023, 16(11): 1733-1742. |
| [25] | Campos LRS, Trefflich S, Morais DAA, Imparato DO, Chagas VS, Albanus RD, Dalmolin RJS, Castro MAA. Bridge: a new algorithm for rooting orthologous genes in large-scale evolutionary analyses. Mol Biol Evol, 2024, 41(2): msae029. |
| [26] | García Mesa JJ, Zhu ZQ, Cartwright RA. COATi: statistical pairwise alignment of protein-coding sequences. Mol Biol Evol, 2024, 41(7): msae117. |
| [27] | Spirin S, Sigorskikh A, Efremov A, Penzar D, Karyagina A. PhyloBench: a benchmark for evaluating phylogenetic programs. Mol Biol Evol, 2024, 41(6): msae084. |
| [28] | Mistry J, Chuguransky S, Williams L, Qureshi M, Salazar GA, Sonnhammer ELL, Tosatto SCE, Paladin L, Raj S, Richardson LJ, Finn RD, Bateman A. Pfam: the protein families database in 2021. Nucleic Acids Res, 2021, 49(D1): D412-D419. |
| [29] |
Song XM, Wang S, Li L. New insights into the regulation of Axin function in canonical Wnt signaling pathway. Protein Cell, 2014, 5(3): 186-193.
doi: 10.1007/s13238-014-0019-2 pmid: 24474204 |
| [30] | Wang YT, Dai GY, Gu ZL, Liu GP, Tang K, Pan YH, Chen YJ, Lin X, Wu N, Chen HS, Feng S, Qiu S, Sun HD, Li Q, Xu C, Mao YN, Zhang YE, Khaitovich P, Wang YL, Liu QX, Han JDJ, Shao Z, Wei G, Xu C, Jing NH, Li HP. Accelerated evolution of an Lhx2 enhancer shapes mammalian social hierarchies. Cell Res, 2020, 30(5): 408-420. |
| [31] |
Liu XM, Fu YX. Exploring population size changes using SNP frequency spectra. Nat Genet, 2015, 47(5): 555-559.
doi: 10.1038/ng.3254 pmid: 25848749 |
| [32] |
Hu WJ, Hao ZQ, Du PY, Di Vincenzo F, Manzi G, Cui JL, Fu YX, Pan YH, Li HP. Genomic inference of a severe human bottleneck during the Early to Middle Pleistocene transition. Science, 2023, 381(6661): 979-984.
doi: 10.1126/science.abq7487 pmid: 37651513 |
| [33] | Muyas F, Sauer CM, Valle-Inclán JE, Li RY, Rahbari R, Mitchell TJ, Hormoz S, Cortés-Ciriano I. De novo detection of somatic mutations in high-throughput single-cell profiling data sets. Nat Biotechnol, 2024, 42(5): 758-767. |
| No related articles found! |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||