早在1866年,“遗传学之父”孟德尔就开始描述和分析表型,在其发表的题为《植物杂交实验》的著名论文中,论述了豌豆的7对相对特征,包括种子的圆形与皱缩、高茎对矮茎、绿色豆荚与黄色豆荚等。随着遗传学的发展,植物学家对植物特征的分析越来越深入。鉴于手工测量的局限性,一些特定的植物特征测量工具逐步兴起,开始进入表型分析的数字化测量阶段。种子、叶片、根系、果实、细胞、植物冠层等不同尺度,都有相应的分析系统或专业软件[5,6,7]。植物某些类型特征的观测深度、测量效率有了实质性的提升。在植物内在特征测定方面,叶绿素荧光技术是研究植物生理过程中应用最广泛的技术之一[8,9]。目前已有许多针对光合作用机理及叶绿素荧光的不同特性而研发的检测设备[8]。这一阶段的特点在于可以弥补手工测量中一些外在特征参数的误差,可无损获取植物内在的特征,获得更多数字化的结果,使得表型分析的研究进程加快。但这些工具只针对单一类型特征进行收集与分析,对于整株所有特征的分析,需要配备不同类型的测量工具,后期操作难度大,数据分析进程缓慢,不适合大规模的遗传群体特征参数的筛选。
直到20世纪末,对于植物表型的采集分析才进入真正意义上的表型组学研究阶段,其核心是获取高质量、可重复的性状数据进而量化分析基因型和环境互作效应(G×E)及其对产量、质量、抗逆等相关的主要性状的影响[10,11]。相对于单一性状分析,植物表型组能为植物研究提供全面的科学证据[12],当与相应的基因组和环境数据相结合时,有望在植物育种领域引发巨大的飞跃[13]。因此,植物科学研究的难点正逐渐从基因分析转向表型分析[14]。表型组学是突破未来作物学研究和应用的关键研究领域,通过表型分析来描述关键性状,可以为育种、栽培和农业实践提供基于大数据的决策支持。另外,表型组学的应用潜力还体现在与其他组学研究的联合分析,如基因组学、表观组学、转录组学、蛋白组学、代谢组学和降解组学等可针对作物的细胞、组织、器官、群体等不同层面以及作物的不同生长发育时期进行综合分析,以绘制各项生命活动过程中的调控网络,最终揭示生命本身的奥秘,揭示农作物的生物学规律,切实服务于农业生产[15]。
推动表型组学研究发展的关键因素之一在于提高表型采集技术及建设表型采集分析设施。Araus等[16]通过比较作物基因组选择和高通量表型组技术后指出,表型获取技术是整个作物育种中比较薄弱的环节,需重点提高该技术的精度及通量。高通量表型组学研究设施的雏形最早是Crop Design公司在1998年研制成功,被定名为Trait Mill平台,已应用于水稻基因及其功能的评价和筛选,并实际应用于作物育种。2008年成立的澳大利亚植物表型组学实验室(Australian Plant Phenomics Facility),总投资超过50 000 000美金,已经成功应用于谷物盐胁迫研究、植物抗旱性研究、抗(硼)毒性研究、高通量谷物生物量精准建模和预测、盐胁迫和根系发育研究等。此后,国际上大的科研机构及跨国种业公司相继建立了高通量的表型分析平台,用于精准采集分析植物表型信息。相对来说,我国的表型组学设施建设起步较晚,可采集分析的表型信息还比较有限。植物表型组学研究平台(Plant Phenomics Analysis Platform, PPAP)于2017年由中国科学院遗传与发育生物学研究所初步建成,是一套包含可见光成像、红外成像、近红外成像、根系近红外成像、荧光 成像、叶绿素荧光成像、高光谱成像和激光雷达 成像8个集成单元的表型研究设施。本文结合国内外植物表型组学研究平台建设与应用方面的进展,以PPAP为基础,从硬件基础建设、各类表型技术开发及服务等方面介绍表型组学研究设施的技术 应用。
1 平台设施建设
由于高通量表型组学技术的兴起,国际上已经在运行的大型表型设施有100多套,最具有代表性的包括澳大利亚国家植物表型设施“植物加速器”、英国国家植物表型中心、德国Julich表型研究中心及德国IPK温室自动传送表型平台等。但我国大型表型分析设施建设仍处于起步阶段,多限于实验室性质的建设工作,用于基因转化、基因分析和鉴定。我国首个大型表型分析平台是华中农业大学与华中科技大学合作研制的一套水稻表型平台,并于2014年开始启用。此后,国内各家单位开始进行表型平台的购置及搭建工作(表1),但总体来说采集通量较小,尤其是用于表型采集的技术单元少,可获取的表型信息有限,很难达到组学分析的高分辨率。相对来说,PPAP基于可控环境下收集主要作物及植物的全方位、多尺度、多维度表型信息,通过与基因型的关联分析,从组学高度系统深入地挖掘基因型-表型-环境三者的内在关系,全面揭示特定生物性状的形成机制。
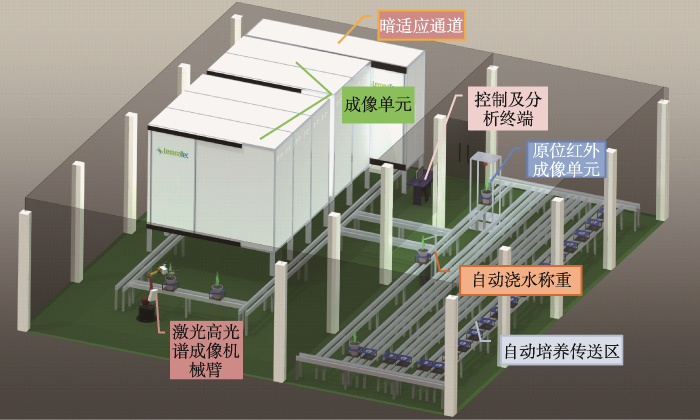
PPAP位于中国科学院遗传与发育生物学研究所西区温室,建设面积为260 m2,由培养区和成像区组成(图1)。其中,培养区包括植物的传送区域、自动称重灌溉设备、环境监测传感器等,用于植物材料的自动化培养和环境信息记录;成像区主要有大型机械臂带动的高光谱和激光雷达成像和基于成像暗室的可见光成像、红外成像、近红外成像、根系近红外成像、荧光成像、叶绿素荧光成像单元。整个设施可供300盆样品进行自动传送测定表型信息,可收集多个光谱谱段的图像信息和光谱信息,同时收集样品的三维点阵信息。
图1
2 技术应用与服务
PPAP在硬件设施建设的基础上,积极构建各种作物的表型采集分析体系,为科研人员提供及时有效的技术支撑。目前该平台已经成功建立了根系可视化及表型分析技术、穗部性状表型分析技术以及抗逆性状表型分析技术等,已为多个研究机构提供各项表型分析服务。
2.1 根系表型分析技术与服务
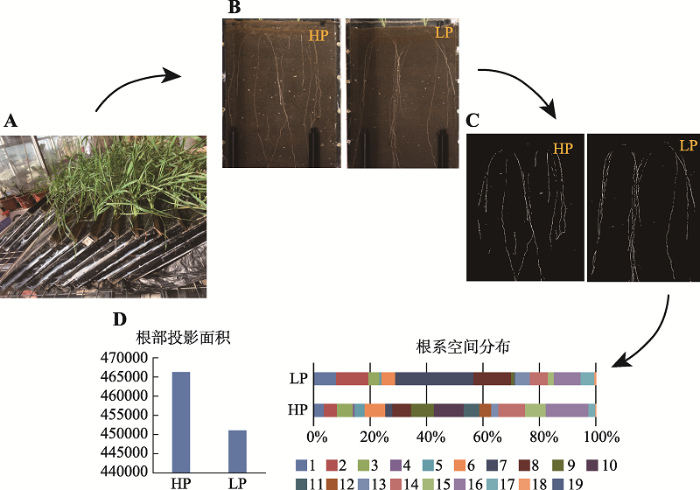
植物根系是植物的重要组成部分,具有非常重要的功能,如水分和养分的吸收与转运、有机物贮藏、植株锚定及与土壤互作等。同时,由于根系生长在地下,根系表型采集的核心在于如何将自然条件生长的根系进行可视化。因此,根系表型性状的采集和分析已成为生物学及表型组学研究的重点和难点。PPAP建立了一种用于高通量植物根系可视化分析的培养及测量技术体系(图2),能够通过根窗技术无损伤、可持续跟踪观测、原位采集土壤根系信息。利用该技术可以对作物根系进行无损伤、高通量和全自动根系表型分析,可测量分析参数如根冠结构(包括跟深和冠幅等)、根冠面积和根长等。
图2
图2
小麦在低磷和高磷不同处理下根系参数的采集分析
A:种植体系;B:图像采集;C:图像处理;D:根系参数提取分析。HP为高磷处理,LP为低磷处理。
Fig. 2
Phenotyping analysis of wheat roots under different concentrations of phosphorus
表1 国内大型植物表型研究设施比较
Table 1
| 平台建设单位 | 成像单元 | 承载通量 | 应用方向 |
|---|---|---|---|
| 中国科学院遗传与发育生物学研究所 | 可见光、近红外、荧光、根系近红外、红外、叶绿素荧光、激光雷达和高光谱 | 300盆 | 小麦、水稻、大豆和玉米等作物的表型分析和相关研究 |
| 中国农业科学院生物技术研究所 | 可见光和近红外 | 456盆 | 玉米和小麦等作物的表型分析和相关研究 |
| 垦丰种业 | 可见光、近红外、荧光和根系近红外 | 680盆 | 玉米和水稻等作物的表型分析和相关研究 |
| 上海乾菲诺公司 | 可见光和近红外 | 252盆 | 全开放平台,承接各种作物和植物表型分析 |
| 中国科学院上海植物逆境研究中心 | 可见光 | 20盆 | 大豆等植物的表型分析 |
| 广西农业科学院甘蔗研究所 | 可见光和近红外 | 15盆 | 甘蔗等热带植物的表型分析 |
| 华中农业大学 华中科技大学 | 可见光、高光谱和电子计算机X射线断层扫描(简称CT) | 2000盆以上 | 水稻和玉米等作物的表型分析和相关研究 |
2.2 穗部性状表型分析技术与服务
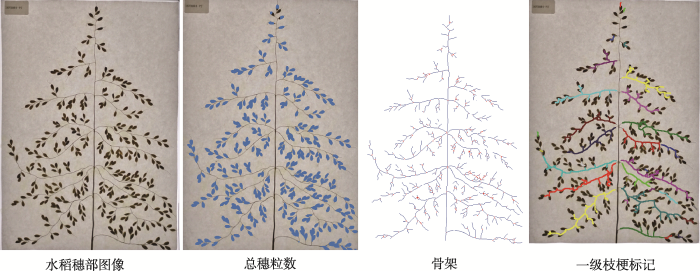
穗部性状是禾谷类作物表型研究的重要部分之一,穗型与作物产量水平和群体结构状况均有着密切关系,因而穗部性状一直是作物育种及栽培研究的焦点,主要包括穗的多少、大小和形态等。PPAP利用可见光的颜色信息和红外成像的温度信息,拟合分析两类图像获得小麦、水稻的单株穗数。通过穗部的扫描图像可以获得穗部组成性状,包括穗型、穗长、每穗粒数、着粒紧密度和枝梗数目等(图3)。
图3
2.3 非生物胁迫表型鉴定分析技术与服务
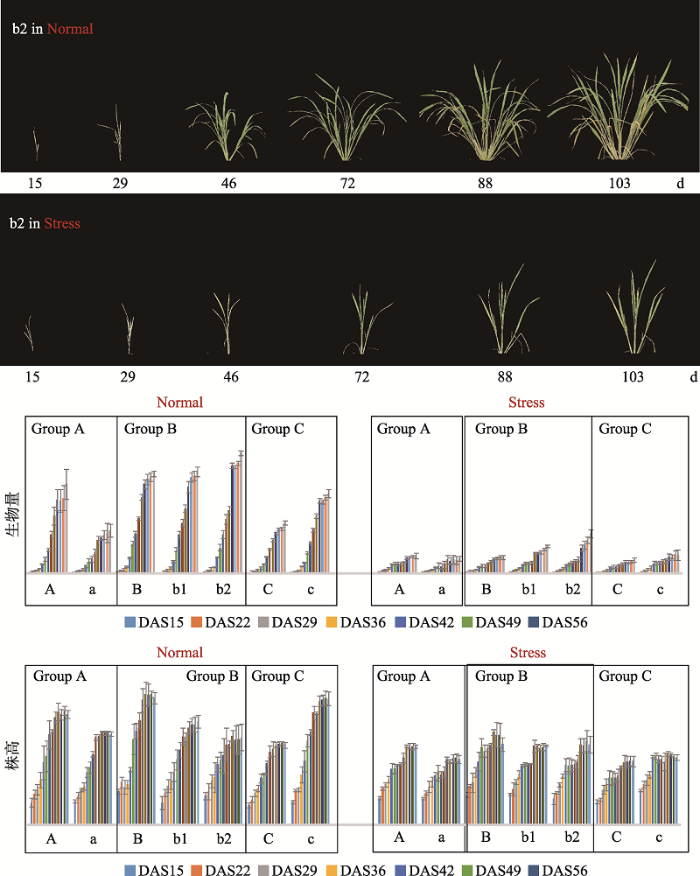
植物对其生存和生长不利的各种环境因素的响应是一个复杂的过程,所有逆境因素包括干旱、高温、低温、盐胁迫和重金属胁迫等逆境造成的植物表型都是复杂性状。对于复杂性状的解析需要获取多维度的表型信息,通过对高分辨率的表型信息的统计分析建模,才有望将非生物胁迫造成的复杂性状解析成可识别的简单指标。PPAP通过采集植物在多个光谱下的光谱反射值,建立特定的图像分析算法流程,可以动态、定量分析植株在干旱、盐胁迫及缺氮条件下表现出的生物量变化、叶片黄化面积、枯萎面积、叶片含水量的动态变化、叶表温度的变化等。通过对这些表型指标的综合分析,建立模型对应到某个逆境胁迫过程植物的表型响应过程。目前,该平台已经开展了小麦和水稻(图4)等作物在干旱、盐胁迫、高温等胁迫过程中的动态表型分析服务。
图4
图4
不同基因型水稻在营养胁迫条件下的生长发育表型检测分析
b2为一个水稻的基因型,Normal表示正常对照处理,Stress表示低氮处理。
Fig. 4
Phenotyping analysis for different genotypes of rice under control and nutrition stress
3 结语与展望
表型组学是突破未来作物学研究和应用的关键研究领域,通过表型采集分析可以为育种、栽培和农业实践提供基于大数据的决策支持。对于表型组学来说,多元数据的组合可以解释新的生物学知识,所以这就要求在表型设施平台建设及数据采集方面应尽量多地考虑多模态数据采集的结合,因为单个传感器获取数据特征是有限的,而且仍然存在一些技术局限性,通过结合多种成像传感器或成像技术可以获得更多的表型特征和更全面的数据集,且不同的成像技术相结合也可以突破单个成像技术的局限性。例如关于叶片生长发育的生物学问题,可以利用可见光成像得到叶片的大小和颜色等信息,再结合多光谱、高光谱和热成像等技术可同时推导出叶片的生化含量,解释叶片的光学特性。目前,植物表型组学研究平台是以光学及光谱成像为主的表型采集手段,主要基于二维图像进行表型的获取和分析。未来会建设规划为整合CT成像技术,可以获得样品的三维及内部结构信息,有效避免光学成像中由于叶片相互遮挡产生的问题,获取更多肉眼不可见的表型信息。该平台预计到2020年底完成CT成像技术与光学成像的结合。
通过多模态成像技术的整合,在技术服务上平台拟通过高光谱成像单元获取的光谱数据及图像数据,建立起植物内在生理表型无损检测分析的技术体系,为科研工作者提供相关技术服务。通过CT成像技术获取的3位信息,建立高通量分析算法,全自动高通量进行表型参数的提供和分析。
表型组大数据的发展,要求表型设施要高通量、多尺度地采集样本的时空表型信息,样品的种养及采集工作也是费时费力的。为推动表型组学的发展,必须促进数据信息的共享使用,其中最重要的就是建立数据标准,通过标准化采集过程、标准化定义描述及标准化数据分析,才能推动表型组大数据的可持续共享。
植物表型组学研究平台是当今植物科学研究发展所需要的重要基础设施,对于推动植物表型组学的发展具有重大意义,例如可以实现量化分析植物个体的全部表现信息,对植物不同空间尺度,如冠层、个体、组织器官甚至细胞及它们在整个生长发育各个阶段进行动态性状的获取和分析。通过与基因组及可控环境数据的关联分析,预期可以绘制各项生命活动过程中的调控网络,最终揭示生命本身的奥秘,揭示农作物的生长发育规律,切实服务于农业生产。
参考文献
The isolation of biochemically deficient mutants of bacteria by means of penicillin
What exactly are genomes, genotypes and phenotypes? And what about phenomes?
Ability of modified spectral reflectance indices for estimating growth and photosynthetic efficiency of wheat under saline field conditions
Molecular distribution and toxicity assessment of praseodymium by
Effects of sodium bicarbonate concentration on growth, photosynthesis, and carbonic anhydrase activity of macroalgae Gracilariopsis lemaneiformis, Gracilaria vermiculophylla, and Gracilaria chouae(Gracilariales, Rhodophyta)
Molecular breeding in developing countries: challenges and perspectives
Breeding technologies to increase crop production in a changing world
With 'phenomics,' plant scientists hope to shift breeding into overdrive
The adoption of automated phenotyping by plant breeders
High-throughput phenotyping for crop improvement in the genomics era
Plant phenomics: history, present status and challenges
植物表型组学: 发展、现状与挑战
Translating high-throughput phenotyping into genetic gain